DNAの多様性:
多型(たけい)ともいうDNAの違いがみられる何がわかるのだろうか.イネの在来種の違い,品種識別,DNAならコメも個体の識別ができます.そのためにDNA抽出と多型の比較が必要であり,その方法には生物種によって使える手段が限られてきました.しかし,現在はあとで触れる次世代シークエンサーやMIG-seq(Suyama and Matsuki 2015:Scientific Reports 5:16963)と呼ばれる方法があるため,コストとの兼ね合いで選択できる方法が増えてきました.
イネでは一塩基多型(これはコスト高くなり,必要な調査機材が多くなります),
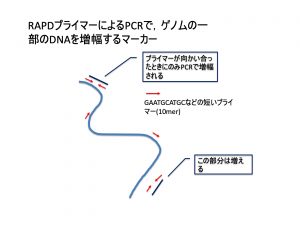
RAPD(ランダムプライマーによるPCR産物によるマーカーシステム),
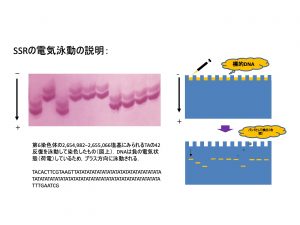
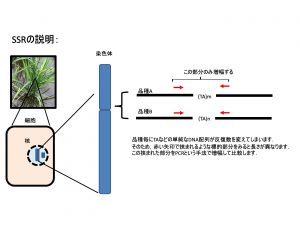
SSR(単純塩基反復配列)マーカー
が使えます.
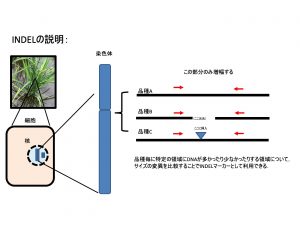
いまはPCR法が利用できるためINDELマーカーの開発もできます.
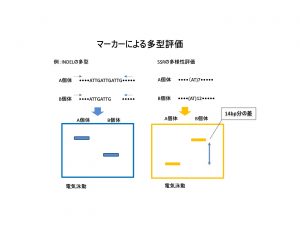
PCRによる増幅後は電気泳動を行ってバンドの識別を行えます.


こちらは在来赤米の多様性を調査したときのものです.
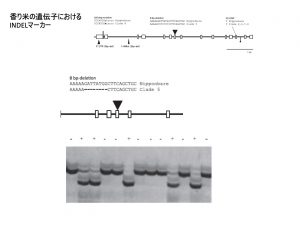
当研究室では香り米も利用することが多いのでINDELマーカーを利用して判定しています.
野生イネ集団解析ではCAPSマーカー開発も行いました.
調査集団(Google)
調査地の現場写真
近づくにはカヌーを利用しました.その結果,
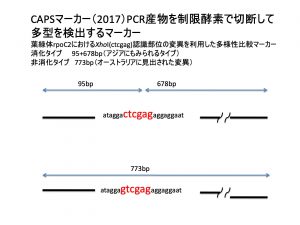
葉緑体の母系情報を得るためにゲノム情報から制限酵素で切断できる場所を選定して,PCR増幅後に消化して電気泳動を行いました.
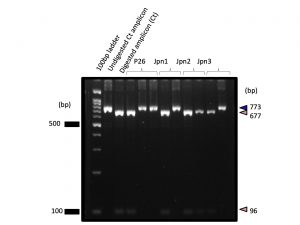
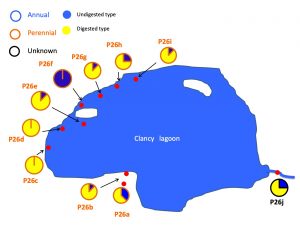
生態特性を含めて1つの池の周辺の集団の多様性が浮かび上がってきました.
*PCRによるマーカー:RAPD,SSR,CAPSなど.利点:少量の試料でよい,DNA多型検出頻度も高いことです.
RAPD:10merのプライマーで数多くのゲノム上で検出される部分を同時に検出
SSR:ゲノム配列の必要性.共優性.近縁品種間でも多型期待できる.染色体のあらゆるところに散在.さらに密なマーカーはSNP.http://archive.gramene.org/markers/microsat/
SNP:一塩基多型.PCRにより特殊なタイピングにより多型をみることが可能.シークエンス情報から位置情報を明らかにしてから用いる.
CAPS:PCR産物内の制限酵素多型を検出する
イネ在来種の場合は,農水省のコアコレクションにおけるSSR多型情報の開示もなされています.